
发布时间: 2025-09-15 16:05:09
最近发表在《J Transl Med》(IF=7.5)的研究,通过多组学整合+机器学习,从1800+差异基因中筛出6大关键基因,其中IRF1、GBP5和PARP9的诊断AUC值高达0.95!更发现它们与M1巨噬细胞浸润、干扰素通路密切相关,甚至通过遗传学验证了IRF1与炎症性肠病(IBD)风险的因果关联!
想了解这些基因如何成为IBD精准诊疗的“密码”?快往下看:
亮点预告:
✨ 机器学习模型横扫113种算法,RF+NaiveBayes组合夺冠;
✨ 免疫浸润分析揭露M1巨噬细胞的“幕后推手”角色;
✨ 首次用孟德尔随机化证实IRF1升高直接增加IBD风险。
题目:利用生物信息学和机器学习识别作为IBD诊断生物标志物的关键基因
杂志:J Transl Med
影响因子:IF=7.5
发表时间:2025.07.03
研究背景
IBD的发病机制复杂,临床缓解仍具挑战性。本研究旨在鉴定IBD的潜在生物标志物,分析其与免疫细胞浸润的相关性,并鉴定与IBD存在因果关系的基因。
数据来源
从GEO检索IBD的RNA测序数据集,分为发现队列(GSE75214)、验证队列(GSE36807)和独立测试队列(GSE179285、GSE47908)。
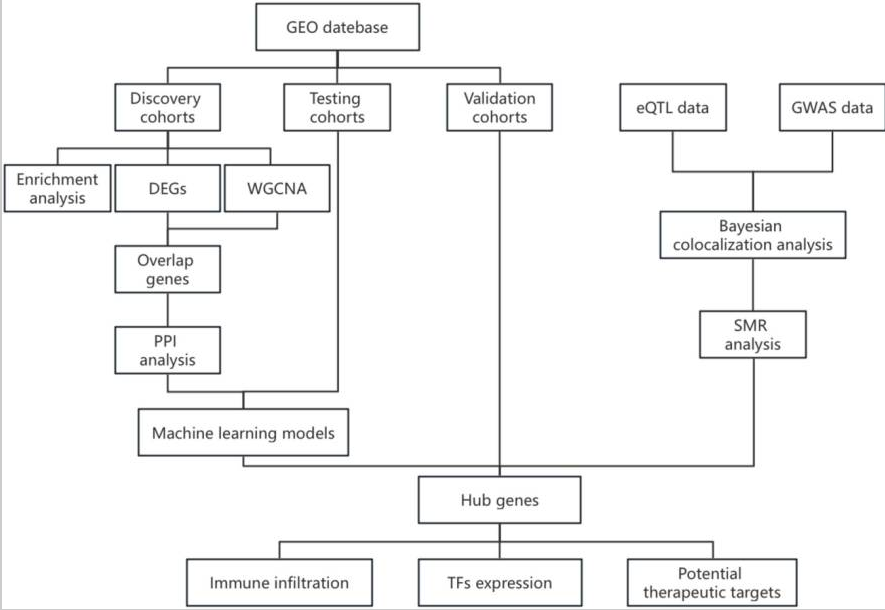
研究思路
通过对发现队列进行比较表达谱分析,检测到了与IBD相关的差异表达基因(DEG)。随后使用PPI分析确定核心候选基因的优先顺序,并通过机器学习方法进一步细化。通过CIBERSORTx反卷积算法进行免疫细胞丰度量化和与IBD相关转录本的统计相关性分析。为了补充这些结果,将来自GTExv8.ALL.Whole_Blood的血液表达数量性状位点(eQTL)数据与来自FinnGen联盟的IBD全基因组关联统计数据整合在一起。该多组学整合框架采用:(1)贝叶斯共定位来评估共享的因果变异,(2)HEIDI异质性检验,以及(3)汇总孟德尔随机化(SMR)来验证因果推断。
主要结果
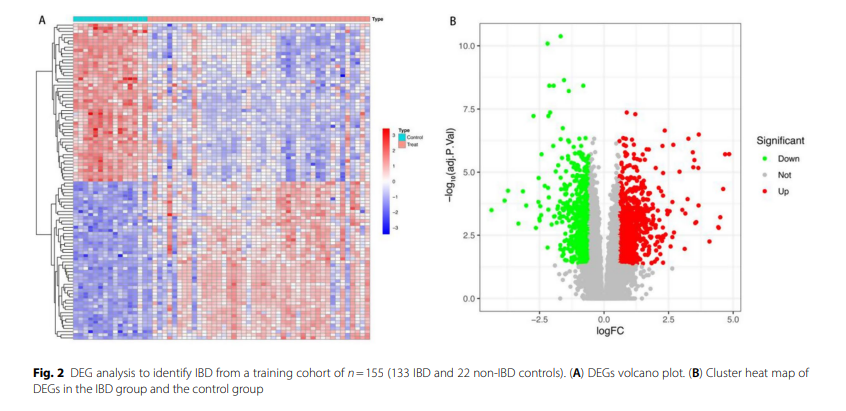
1、差异基因鉴定
IBD组与对照组之间共鉴定出1816个DEG,log2FC>0.585,FDR<0.05。
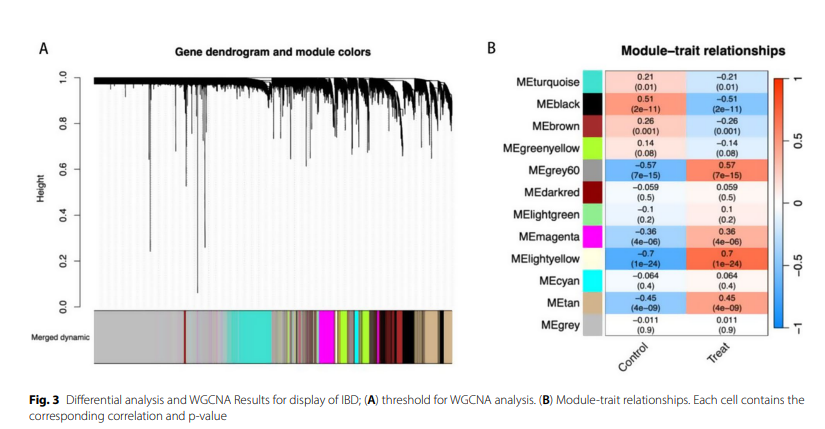
2、WGCNA确定了与IBD密切相关的DEG上的关键模块
MElightyellow、MEblack和MEgrey60与IBD呈显著正相关,且各模块间基因显著性(GS)分布存在显著差异。随后进行模块内分析,结果显示MElightyellow中模块成员(MM)与基因显著性(GS)呈显著正相关。最后,将MElightyellow模块中的基因作为IBD相关基因进行进一步分析。
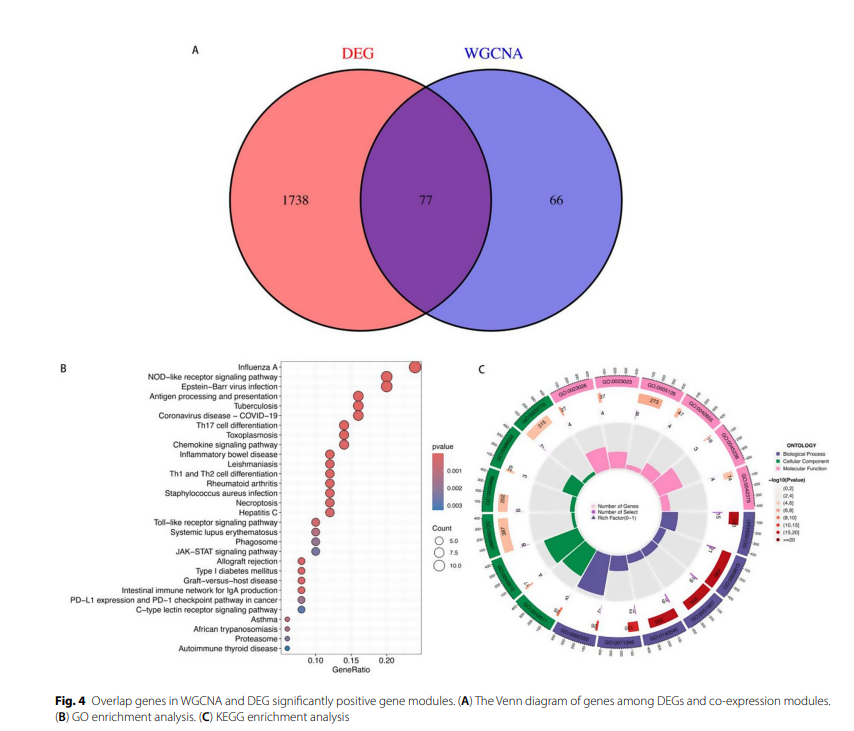
3、关键模块之间的大量重叠表明与IBD相关的生物学功能和通路之间存在密切的关系
比较WGCNA和DEG显著阳性基因模块,得到这些已鉴定模块的交集。随后,对交集基因进行富集分析。GO富集分析表明,77个基因与生物膜高度相关。KEGG富集分析发现,77个基因主要与甲型流感病毒和EB病毒感染相关。
4、通过PPI网络分析与机器学习的整合筛选,获得了6个IBD特征基因
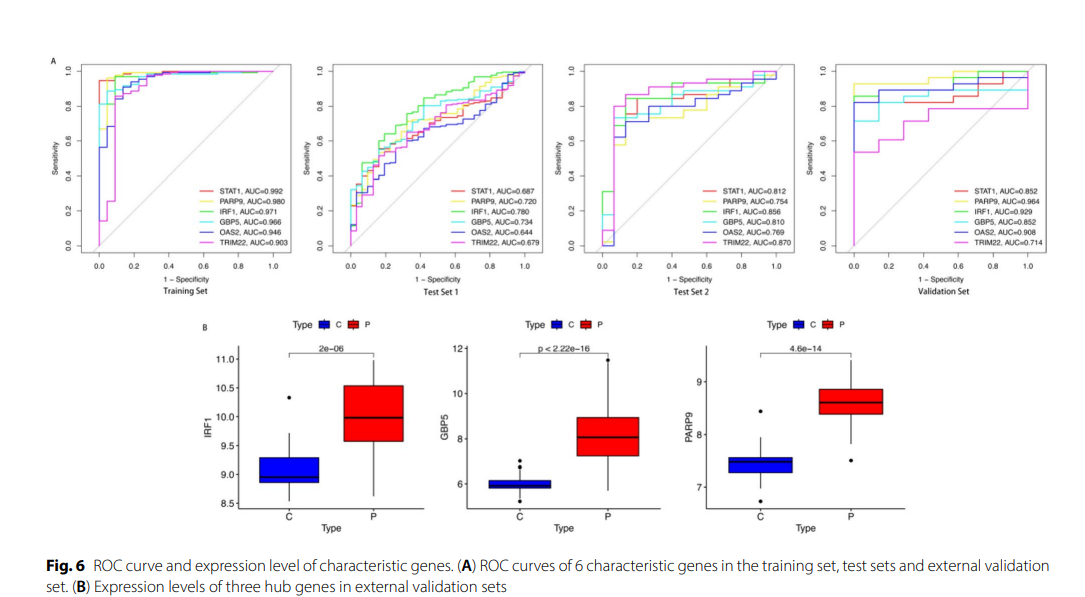
利用cytoHubba中的Degree算法,生成PPI的交互网络并识别出15个最重要的关键基因。基于机器学习整合15个基因表达谱,筛选IBD关键基因。本研究构建了113个预测分类模型,RF+NaiveBayes整合算法在训练集和测试集中均获得了最高的平均AUC得分,被认定为最有效的分类模型。最终,通过RF+NaiveBayes算法,获得了一组6个特征基因:STAT1、PARP9、IRF1、GBP5、OAS2和TRIM22。
5、IRF1、GBP5和PARP9是IBD的诊断生物标志物
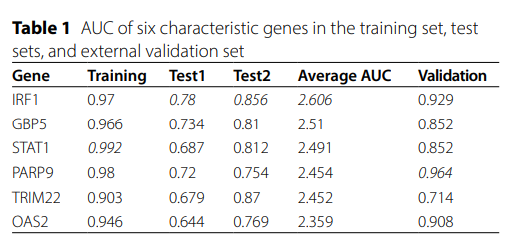
为了评估这六个特征基因的诊断能力,我们在训练组和测试组分别进行了ROC曲线分析。结果显示,在训练组中,IRF1、GBP5和PARP9的AUC值均大于0.95。因此,IRF1、GBP5和PARP9被认定为IBD的中心基因,可以作为潜在的诊断标志物。此外,基于这三个中心基因的组合构建了模型,结果显示,在训练组和测试组中,这三个中心基因的诊断性能均显著提高。在三个不同的数据集中评估了IRF1、GBP5和PARP9的诊断准确性、相关性和表达模式(表1)。
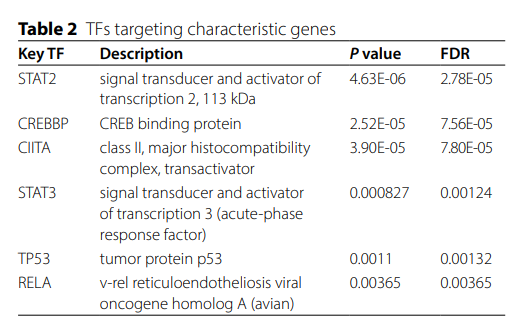
6、CIITA、STAT3、RELA、STAT2是针对特征基因IRF1和STAT1进行主动靶向的转录因子(TF)
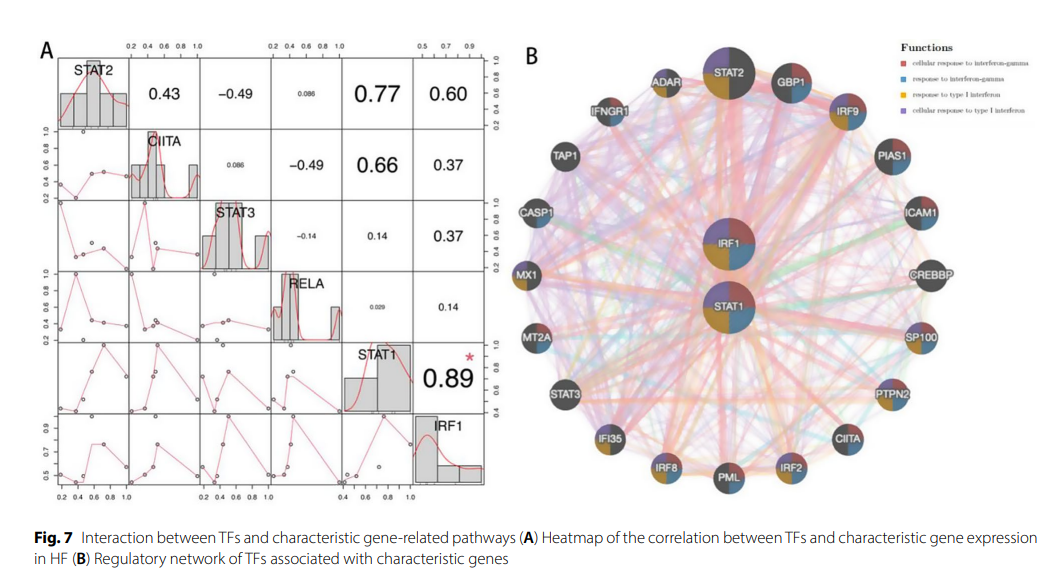
基于TRRUSTv2数据库,共招募到6个针对特征基因的TFs(STAT2、CREBBP、CIITA、STAT3、TP53、RELA)。在训练集和测试集中,共有4个TFs(CIITA、STAT3、RELA、STAT2)在IBD中表达上调,被认为是IBD进展的关键TFs。上述TFs均与IRF1和STAT1相关。图7(A)进一步展示了2个特征基因和4个关键TFs在IBD中的关联模式,其中STAT2与IRF1和STAT1显著相关。IRF1和STAT1相关TFs的调控网络如图7(B)所示。富集功能排名前三的分别是细胞对干扰素-γ的应答、对干扰素-γ的应答、以及对I型干扰素的应答。
7、巨噬细胞M1细胞浸润增高和浆细胞浸润减少与中心基因高度相关
在IBD训练组和验证组中,M1巨噬细胞的浸润均显著增加,并与中心基因呈正相关,这表明M1巨噬细胞和浆细胞在由中心基因调控的免疫相关通路中发挥关键作用。
8、IBD患者IRF1升高具有很强的诊断价值,并且与IBD风险呈正相关
为了探究与遗传变异相关的基因生物标志物的潜力,基于eQTL数据和大规模GWAS数据进行了SMR分析,随后进行了HEIDI分析。经过Bonferroni校正和共定位分析,发现133个基因与IBD风险相关。其中,IRF1也是IBD的核心基因。最终发现,较高的IRF1可能对应于IBD风险的增加。
文章小结
已鉴定出多个与IBD相关的潜在基因,包括IRF1、GBP5和PARP9。基于eQTL数据的SMR分析显示,IRF1与IBD风险显著相关。需要进一步开展功能研究以阐明相关机制,这将有助于推动IBD靶向分子治疗和免疫疗法的发展。





上一篇:缺B12,不止是贫血:肺纤维化与哮喘的潜在危险因子?