
发布时间: 2024-02-28 16:10:30
2024年1月,最新刊登于《Journal of Translational Medicine》的《Gastroesophageal Reflux Disease and the Risk of Respiratory Diseases: A Mendelian Randomization Study》引起了我们对呼吸系统健康的极大兴趣。这项由中国学者完成的研究,其高达7.4的影响因子证实了其在医学领域的重要性。
在这篇研究中,作者运用先进的孟德尔随机化方法,通过基因组关联研究数据深入研究了GERD与呼吸系统疾病的潜在因果关系。结果表明,患有胃食管反流病(GERD)的人患慢性阻塞性肺病、支气管炎、肺炎、肺癌和肺栓塞的风险更高。
孟德尔随机化,Mendilian Randomization,简写为MR,是一种在流行病学领域应用广泛的一种实验设计方法,利用公开数据库就能轻装上阵写文章,甚至掌握发表高质量的论文的关键。
孟德尔随机化通过引入一个称之为工具变量的中间变量,来分析暴露因素和结局之间的因果关系,解决了传统实验方法由于混杂因素的存在,而无法有效说明暴露因素和结局变量之间因果性的问题。想象一下,我们希望了GERD是否与呼吸系统疾病的发生有关。在MR中,我们可以使用基因作为 '患有GERD' 和 '不患有GERD' 的二元变量。这种方法允许我们通过观察基因变异对GERD的影响,全面评估患GERD对呼吸系统疾病的潜在因果效应。通过阅读本文,您将掌握开展相似研究的关键方法和技巧。

1 摘要与主要研究
1.1 摘要
背景: 观察性研究已经暗示胃食管反流病(GERD)与呼吸道疾病存在关联,但因果关系仍然存在争议。本研究旨在通过采用孟德尔随机化(MR)研究评估GERD在呼吸道疾病中的潜在因果作用。
方法: 我们利用全基因组关联研究(GWAS)的摘要数据进行了孟德尔随机化分析,采用三种MR分析方法(IVW、WM和MR-Egger)来评估GERD与呼吸道疾病风险之间的可能因果关系。为确保更可靠的结果,我们进行了敏感性分析,包括检查异质性、多效性以及留一法分析。同时,我们鉴定了33个相关基因,并研究了它们在26个正常组织中的分布。
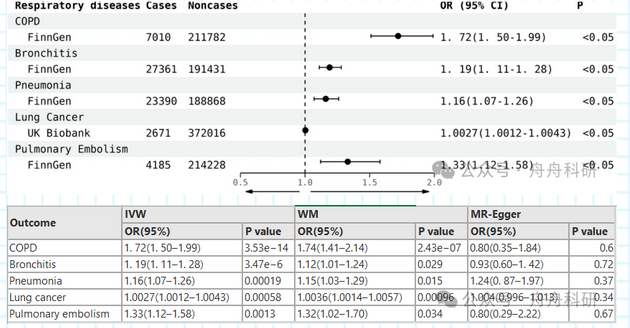
结果: 在分析中,GERD发病率每增加一个单位,患慢性阻塞性肺疾病(COPD)、支气管炎、肺炎、肺癌和肺栓塞的几率分别增加了72%(OR(IVW)=1.72,95%置信区间:1.50~1.99)、19%(OR(IVW)=1.19,95%置信区间:1.11~1.28)、16%(OR(IVW)=1.16,95%置信区间:1.07~1.26)、0.3% (OR(IVW)=1.003,95%置信区间:1.0012~1.0043)和33%(OR(IVW)=1.33,95%置信区间:1.12~1.58)。此外,研究中未观察到明显的异质性和多效性。该研究还发现,这些基因在中枢神经系统和脑组织中的表达水平高于其他正常组织。
结论: 本研究提供证据表明,患GERD的个体患慢性阻塞性肺疾病(COPD)、支气管炎、肺炎、肺癌和肺栓塞的风险较高。我们的研究建议医生对GERD患者进行有效的呼吸道疾病治疗。通过对基因表达的探索,我们的研究还可能有助于揭示中枢神经系统和脑组织在GERD引发的呼吸道疾病中所扮演的角色。
1.2 研究结果
1.2.1 GERD对呼吸道疾病的因果影响
通过使用逆方差加权法,我们确定了GERD和呼吸道疾病之间的因果关系。我们观察到,基因预测的GERD与呼吸道疾病的风险呈正相关(COPD:OR(IVW)=1.72,95%置信区间:1.50~1.99;支气管炎:OR(IVW)=1.19,95%置信区间:1.11~1.28;肺炎:OR(IVW)=1.16,95%置信区间:1.07~1.26;肺癌:OR(IVW)1.003,95%置信区间:1.0012~1.0043;和肺栓塞:OR(IVW)=1.33,95%置信区间:1.12~1.58)。
1.2.2 敏感性分析
在敏感性分析中,我们进行了MR-Egger回归,发现COPD、支气管炎、肺炎、肺癌和肺栓塞的水平多效性证据有限(分别为P = 0.50、0.94、0.89、0.73和0.57)。此外,MR-PRESSO方法未发现离群点,表明水平多效性可能性非常弱。为了评估COPD、支气管炎、肺炎、肺癌和肺栓塞的异质性,我们采用了Cochrane's Q检验,这可以帮助我们判断不同基因之间是否存在异质性,检验结果展示所有P值均>0.05(分别为P = 0.07、P = 0.25、P = 0.78、P = 0.37和P = 0.32)。
1.2.3 MERAV 数据库分析
MERAV数据库分析显示,相关基因在中枢神经系统和脑组织中的表达水平较高。
2 统计学方法
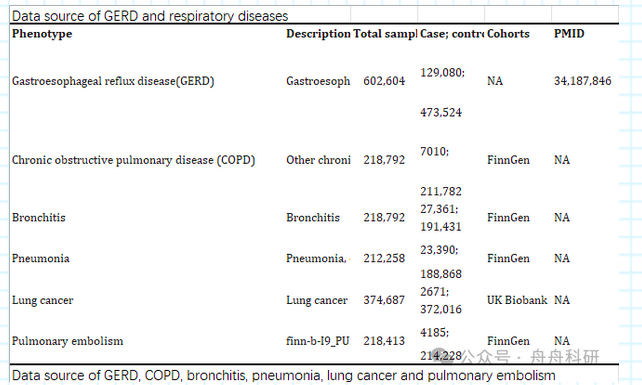
2.1 GERD的数据来源
GERD的GWAS数据来源于综合流行病学单位(IEU)的公开摘要数据(https://gwas.mrcieu.ac.uk/),研究总共分析了129,080例GERD病例和473,524例对照。COPD、支气管炎、肺炎、肺癌和肺栓塞的汇总统计数据来自FinnGen和UK Biobank的公共数据库。为了减少人口分层偏倚的风险,所有数据均来自欧洲人群。
2.2单核苷酸多态性选择
将去除连锁不平衡的阈值设置为r2<0.001,全基因组显著性阈值设置为P = 5× 10−8后,我们计算F统计量来量化SNP的强度,并随后消除F统计量小于10的SNP。
然后,我们使用效应等位基因频率来协调相应的暴露和结局数据集。数据来源于dbSNP的ALFA等位基因频率[Home-SNP-NCBI (https://www.nih.gov/)]。
2.3 孟德尔随机化
使用随机效应逆方差加权(IVW)方法进行了主要研究,同时利用加权中位数和MR-Egger方法进行了辅助分析。对SNP特定的Wald比率估计进行元分析,即SNP对结果影响的β系数除以SNP对暴露影响的β系数,使用随机效应或固定效应逆方差方法对每个比率进行加权(按其标准误差加权),用于计算因果估计。加权中位数方法计算了SNP估计之间的加权中位数。
在本研究中,使用Cochran’s Q检验的P值来评估分析中的异质性,当Cochran’s Q检验的P ≥ 0.05时,认为因果分析中不存在异质性。采用漏斗图检测异质性。我们用MR-PRESSO来评估模型的多效性,消除了导致偏倚的SNP。通过MR-Egger回归分析,可以评估水平多效性引起的偏倚,并利用回归截距计算多效性的大小。此外还使用了留一法进行敏感性分析。
2.4 组织水平SNP遗传丰度
在这项研究中,我们通过搜索dbSNP中的关键SNP来鉴定基因(https://www.ncbi.nlm.nih.gov/snp/)。然后,我们在代谢基因快速可视化数据库(MERAV, http://merav.wi.mit.edu)中进行数据挖掘,探索33个基因在26个正常组织中的分布。
小结
本文详细内容可在《Journal of Translational Medicine》找到,文章的DOI为10.1186/s12967-023-04786-0。您也可以点击原文阅读获取全文,我们期待您从这项研究中获得有益的信息。这项研究表明,患有胃食管反流病(GERD)的人患慢性阻塞性肺病、支气管炎、肺炎、肺癌和肺栓塞的风险更高。如果您对本研究的统计方法产生兴趣,我们推荐查看在线孟德尔随机化词典(https://mr-dictionary.mrcieu.ac.uk/),以帮助您更深入学习统计方法。
下一篇:我出“故事”,你出“酒”,让我们一起来看这篇7分plus的肿瘤+GEO数据库挖掘是如何娓娓道来的